Betrachtungen über eine Faktoranalyse
auf den SGIPT-Seiten zur statistischen Methodik
Quelle: Siehe http://www.sgipt.org/wisms/fa/Quader/q02.htm (1)
Verwendeter Datensatz: Siehe unten.
Motivation und Kontext
Dieses Posting meinerseits in der Mathematik-Newsgruppe beschreibt den Auslöser für meine besondere Aufmerksamkeit, da der Autor der Seite dort eine PCA auf der Basis von Interaktions-Termen diskutiert um damit die Akkuratheit der Reproduktion einer Korrelationsmatrix durch (eine geringe Zahl von) Hauptkomponenten (hier: 3 von 8) zu illustrieren - ein für mich überraschendes Herangehen...
|
|
Posting in news://de.sci.mathematik und news://de.sci.psychologie 30.10.2014: |
|
|
|
|
Ich habe mich mal wieder auf den Faktoranalyse-Seiten von Rudolf (SGIPT: http://www.sgipt.org ) umgesehen und dort in
http://www.sgipt.org/wisms/fa/Quader/q00.htm
ein merkwürdiges Beispiel gefunden, mit dem die Qualität der Reproduktion einer Korrelationsmatrix via Faktoranalyse nach Hinzufügen eines gewissen Maßes an Fehlerrauschen illustriert werden soll.
Es wird hier ein Beispiel verwendet, in dem auf der Basis von 3 "unabhängigen" Zufallsgrößen (also den impliziten und aufzudeckenden Faktoren) 5 abhängige Größen berechnet worden sind, aus deren Korrelationsmatrix (in einer echten) Faktoranalyse dann also die unabhängigen Faktoren wieder identifiziert/sichtbar gemacht werden sollen. In diesem Beispiel geht es nun zwar lediglich darum, ein Gefühl dafür zu bekommen, wie gut die Korrelationen reproduziert werden, wenn ich nach der Faktoranalyse (genauer genommen PCA) nur die 3 gefundenen relevanten Hauptkomponenten verwende und außerdem in dieses System Fehlergrößen verschiedenen Ausmaßes einstreue.
Das Beispiel als solches erscheint mir allerdings für die Beschreibung der Vorgänge der FA/PCA etwas seltsam gewählt: während die FA/PCA ja eigentlich dafür konzipiert ist, additive Kompositionen aufzudecken, also z.B.
x1 = a1*f1 + b1*f2 x2 = a2*f1 + b2*f2 x3 = a3*f1 + b3*f2
wobei f1,f2 die unbekannten Werte der zugrundeliegenden Faktoren, und x1,x2,x3,... die Werte der "empirisch" beobachteten, durch die unbekannten Faktorenwerte zusammengesetzten, Variablen enthalten, wird hier mit multiplikativen* Kompositionen gearbeitet, z.B.
x1 = f1*f2 x2 = f1*f3 x3 = f2*f3 x4 = f1*f2*f3
wobei f1=länge, f2=breite, f3 = Höhe
als Zufallswerte synthetisch erzeugt werden und in einer echten Faktorenanalyse als Faktoren durch die Messungen von Volumen, Oberfläche etc wiedergefunden werden sollen.
Eigentlich wird hier also die Faktorenanalyse auf das gestützt, was wir in Regression und Anova-Modellen als "Interaktionsterme" bezeichnen; diese Art von Modell für die FA/PCA, und insbesondere die vollständige Modellierung durch diese Interaktionsterme erscheint mir aber als sehr ungewöhnlich, jedenfalls habe ich in allen meinen Arbeiten damit solche Modelle noch nie betrachtet. Interaktionsterme sind zwar in der Regression und in der Anova relativ üblich (z.B. in SPSS einfach zu modellieren) -wenn auch eher als zusätzliche Option- , aber als Items in der FA/PCA ? Das würde mich jetzt einmal interessieren.
Hat jemand hier mehr Erfahrung damit ? (just curious!)
Gottfried
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
Anhang: Ich habe mal neugierigerweise dieses Modell "linearisiert", indem ich die berechneten Werte für die x1,x2,x3,... logarithmiert habe und somit die Produkte in Summen (wie von der FA/PCA erwartet) verwandelt.
Da dies auch bedeutet, die Daten als ratio-skaliert zu verstehen (die Position des Nullpunkt ist nicht trivial) habe ich die Daten dann auf den Nullpunkt zentriert indem ich einfach den Datensatz verlängert habe mit den jeweils negativen Werten der "Messungen" in x1,x2,.... so daß für jede Variable der Mittelwert auf Null geht.
Mit solcherart transformierten Werten erhalte ich dann
* überraschenderweise sehr hohe Korrelationen der Items untereinander,
aber nichtsdestotrotz via multipler Regression
* eine perfekte Reproduktion der zugrundeliegenden Komposition über die unstandardisierten beta-Gewichte
Ergebnisse:
* Regressionsmodell (via PCA über Kovarianzmatrix, Rohdaten zu Logarithmen transformiert):
unstandardisierte beta-Werte * Dependent items (as compositions of the independents): x1=Vol 1.000 1.000 1.000 keine Residualvarianz x2=Ofl 0.799 0.740 0.993 eine kleine Menge Residualvarianz x3=L*B 1.000 1.000 0.000 keine Residualvarianz x4=L*H 1.000 0.000 1.000 keine Residualvarianz x5=B*H 0.000 1.000 1.000 keine Residualvarianz -------------------------------------------------------------------------------------- * Independent items (explaining the dependent items): f1=L 1. 0. 0. f2=B 0. 1. 0. f3=H 0. 0. 1.
Aber dies nur am Rande.
Übrigens hat dann - als einzige- die Variable x2 (="Ofl") einen weiteren (kleinen) Varianzanteil da hier die Logarithmierung einer Summe Ofl = 2∙(f1 ∙ f2+f2 ∙ f3 + ...) eine zusätzliche (wenn auch kleine) Nichtlinearität hinzubringt.
|
|
|
|
|
|
|
|
Kennwerte (aus dem angegebenen Datensatz reproduziert)
Der angegebene
Datensatz (Kopie siehe Ende des Dokuments) ist das zweite Beispiel (...q02)
mit eingestreuten Fehlern unter http://www.sgipt.org/wisms/fa/Quader/q02.htm
Korrelationsmatrix:
|
Neu berechnet aus angegebenen Rohdaten |
Dokumentiert in (1) |
|||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
Die aus den dokumentierten Rohdaten berechnete Korrelationsmatrix ist positiv definit mit einem kleinsten Eigenwert weit genug von dem Maschinen-epsilon entfernt, so daß auch die Cholesky-Dekomposition problemlos mit genügend Stellen Genauigkeit möglich ist.
|
Original data with 17, input read with 17, computet with 19, and showed with 3 digit accuracy (for control here the analysed original matrix):
Läng Breit Höhe Volum Ober L*B L*H B*H 1 -.099 .102 .551 .566 .632 .721 .053 -.099 1 .122 .478 .542 .614 .057 .712 .102 .122 1 .608 .663 .211 .704 .704 .551 .478 .608 1 .981 .843 .823 .779 .566 .542 .663 .981 1 .849 .846 .82 .632 .614 .211 .843 .849 1 .585 .545 .721 .057 .704 .823 .846 .585 1 .488 .053 .712 .704 .779 .82 .545 .488 1
The matrix is not positive definit. Cholesky decomposition is not success-
|
|||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
|
Anmerkung: es ist überraschend, daß es überhaupt möglich sein soll, aus dem Produkt des (z-standardisierten) Rohdatensatzes Z mit sich selbst überhaupt eine nicht positiv-semidefinite Korrelationsmatrix zu erzeugen. Hier müssen gravierende Rundungsfehler unterlaufen sein! |
Eigenwerte:
|
|
Wert |
Log10(wert) |
|
dokumentiert in (1) |
|
Ev1 |
5.04807735 |
0.70312600 |
|
5.08905 |
|
Ev2 |
1.52701773 |
0.18384408 |
|
1.53238 |
|
Ev3 |
1.13576922 |
0.05529010 |
|
1.1371_ |
|
Ev4 |
0.11533442 |
-0.93804107 |
|
.1064_ |
|
Ev5 |
0.10071085 |
-0.99692372 |
|
.09161 |
|
Ev6 |
0.06805516 |
-1.16713892 |
|
.05861 |
|
Ev7 |
0.00500776 |
-2.30035672 |
|
-5.07D-3 |
|
Ev8 |
0.00002750 |
-4.56066522 |
|
-.01008 |
Anm.: die Korrelationsmatrix im Original (1) oder die Eigenwertanalyse muß mit großer numerischer Ungenauigkeit gerechnet worden sein: durch die Konstruktion der Berechnungsweise einer Korrelationsmatrix können negative Eigenwerte bei reellen Rohdaten (bei genügender numerischer Genauigkeit beim Rechnen) nicht entstehen.
Cholesky-Ladungsmatrix:
|
|
f1 |
f2 |
f3 |
f4 |
f5 |
f6 |
f7 |
f8 |
|
Diagonaleinträge |
|
L |
1. |
. |
. |
. |
. |
. |
. |
. |
|
1 _ |
|
B |
-0.0983 |
0.9952 |
. |
. |
. |
. |
. |
. |
|
.9951 |
|
H |
0.1007 |
0.1312 |
0.9862 |
. |
. |
. |
. |
. |
|
.9859 |
|
Vol |
0.5455 |
0.5296 |
0.4838 |
0.4336 |
. |
. |
. |
. |
|
.4149 |
|
OFl |
0.5608 |
0.5948 |
0.5289 |
0.2186 |
0.0650 |
. |
. |
. |
|
-.011_ |
|
L∙B |
0.6258 |
0.6726 |
0.0580 |
0.2515 |
0.0585 |
0.2931 |
. |
. |
|
-.6477 |
|
L∙H |
0.7143 |
0.1274 |
0.6169 |
0.1370 |
0.0836 |
-0.0976 |
0.2404 |
. |
|
-.9996 |
|
B∙H |
0.0523 |
0.7137 |
0.6066 |
0.1632 |
0.0186 |
-0.1716 |
-0.2513 |
0.0158 |
|
-1.1287 |
Anm.: betreffs der negativen Diagonaleinträge in (1) siehe Anm. zu Korrelationen und Anm. zu Eigenwerten.
Die ersten 3 Hauptkomponenten:
|
|
hier berechnet |
|
dokumentiert in (1) |
||||
|
|
PC1 |
PC2 |
PC3 |
|
PC1* |
PC2* |
PC3* |
|
L |
0.5613 |
-0.7370 |
-0.3142 |
|
.563 |
-.738 |
-.314 |
|
B |
0.5313 |
0.7243 |
-0.3942 |
|
.533 |
.725 |
-.395 |
|
H |
0.6639 |
0.0634 |
0.7264 |
|
.667 |
.064 |
.727 |
|
Vol |
0.9725 |
-0.0291 |
-0.0271 |
|
.976 |
-.029 |
-.027 |
|
OFl |
0.9996 |
0.0031 |
-0.0086 |
|
1.004 |
3D-3 |
-9D-3 |
|
L∙B |
0.8372 |
-0.0282 |
-0.5063 |
|
.841 |
-.028 |
-.507 |
|
L∙H |
0.8400 |
-0.4456 |
0.2402 |
|
.843 |
-.446 |
.24 |
|
B∙H |
0.8115 |
0.5050 |
0.1979 |
|
.815 |
.506 |
.198 |
Anm.: (*) = dokumentiert in (1)
|
hier berechnet |
dokumentiert in (1) |
|||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
Reproduzierte Korrelationsmatrix basierend auf 3 Hauptkomponenten:
|
Reproduktionsmatrix aus 3 Faktoren:
.961 -.111 .1 .58 .566 .654 .729 .023 -.111 .967 .115 .51 .541 .628 .031 .724 .1 .115 .977 .629 .663 .19 .709 .719 .58 .51 .629 .955 .98 .835 .83 .775 .566 .541 .663 .98 1.007 .848 .843 .818 .654 .628 .19 .835 .848 .964 .6 .57 .729 .031 .709 .83 .843 .6 .968 .509 .023 .724 .719 .775 .818 .57 .509 .959
|
|||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
Fehler:
|
Residualanalyse Original-Korrelationen und aus 3 Faktoren reproduzierte: .039 .012 1D-3 .029 0 .022 8D-3 .029 .012 .033 7D-3 .032 1D-3 .014 .026 .011 1D-3 7D-3 .023 .022 0 .02 4D-3 .015 .029 .032 .022 .045 1D-3 8D-3 7D-3 3D-3 0 1D-3 0 1D-3 7D-3 1D-3 3D-3 2D-3 .022 .014 .02 8D-3 1D-3 .036 .015 .025 8D-3 .026 4D-3 7D-3 3D-3 .015 .032 .021 .029 .011 .015 3D-3 2D-3 .025 .021 .041 |
|||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
|
Anm.: in der Residualtabelle sollte der rot-markierte Wert negativ sein; das Softwareprogramm scheint hier Fehler zu haben oder einfach alle Residuen als absolute Werte zu dokumentieren. |
Statistiken über wiederholte Zufallsexperimente mit hinzugefügten Fehlern
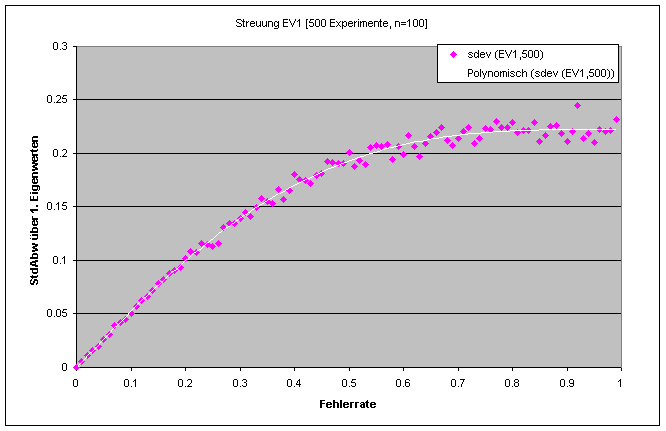
Mit Fehlerraten a zwischen 0 und 2 (als relativer normalverteilter Fehler den Rohdaten hinzugefügt) in Schritten von 0.1 wurden mit jeder Fehlerrate 50 Experimente mit (jeweils neuen) Zufallsfehlern durchgeführt.
Die durchschnittlichen Größen der jeweiligen ersten Eigenwerte sowie deren Streuungen werden im folgenden grafisch dokumentiert.
Die kontinuierlichen Linien entstehen durch kubische Interpolationssplines der Software Excel. Für die durchschnittlichen Werte der EV1 wurden außerdem die 95% Konfidenzintervalle für die Inferenz auf eine ideale Population von Versuchen ausgewiesen (hier ist simpel der SEMean verwendet worden, möglicherweise empfiehlt sich in einer genaueren Untersuchung ein besserer Streuungskoeffizient!).
Berechnungsmodus für einen Datenpunkt xa,r,c: mit der Fehlerrate a: xa,r,c = xr,c ∙(1+ a∙Normal(0,1)) wobei r den Zeilenindex und c den Spaltenindex der Datentabelle bezeichnen.
|
|
|
|
|
|
Anm.: Fehlerraten von 1 (und höher) heißt hier, daß die Originaldaten durch Fehler derselben Größenordnung wie sie selbst überlagert werden; die entsprechenden Korrelationsmatrizen und die Eigenwertverläufe sollten sich deshalb denen von reinen Zufallsdaten annähern. Eine weitere Steigerung der Streuung der jeweils ersten Eigenwerte (bei hier 50 Sample-Ziehungen) ist ab einer relativen Fehlerrate von a>1 oder anscheinend bereits bei a>0.6 deshalb nicht zu erwarten. Deutlicher wird das in den Grafiken für 500 Ziehungen pro Fehlerrate, weshalb hier die x-Achse auf das Intervall 0..1 gekürzt werden konnte.
Gottfried Helms, 31.10.2014
Daten
Die Daten entstehen durch einen Zufallsdatensatz der drei "unabhängigen" Items Länge, Breite, Höhe jeweils zwischen 0 und 100; hiervon abgeleitet sind Vol=L∙B∙H, OFl= 2(L∙B + L∙H + B∙H), und deren Einzelkomponenten L∙B, L∙H, B∙H . Diesen (fixierten) Werten (als konstante Datenbasis für alle Experimente in http://www.sgipt.org/wisms/fa/Quader/q00.htm verfügbar) wird bei jeder "Ziehung" ein relativer Fehler mit der Rate a überlagert. Aus 10, 30, 50 oder mehr Ziehungen mit demselben Parameter a wird dann der durchschnittliche erste Eigenwert berechnet. Soweit ich die Erklärungen in der Webseite verstanden habe, läuft es hierbei auf die Formel
xa,r,c = xr,c∙(1+a∙Normal(0,1)) wobei r den Zeilenindex und c den Spaltenindex der folgenden Tabelle bezeichnen
hinaus, wobei der Zufallsgenerator Normal(0,1)=Normal(mean,stddev) möglicherweise in einer Weise gestutzt wird, daß es wohldefinierte minimale und maximale Werte gibt.
Für die eigene Statistik mit variablen Fehlerraten a wurde der unverzerrte Datensatz herangezogen und für jede "Ziehung" erneut zufällige Fehler darauf gerechnet.
Datensatz in der Version ("...q02" mit Fehlerüberlagerung), kopiert aus Webpage http://www.sgipt.org/wisms/fa/Quader/q02.htm :
|
Lfd. Nr |
L |
B |
H |
Vol |
Ofl |
L∙B |
L∙H |
B∙H |
|
1 |
0.995 |
94.203 |
61.759 |
5839.5 |
11997 |
94 |
62 |
5819.8 |
|
2 |
2 |
65.4 |
70.9 |
9192.8 |
9764 |
130.3 |
141.97 |
4609.5 |
|
3 |
3 |
19 |
39.1 |
2229.6 |
1831.3 |
57.3 |
116.83 |
739.32 |
|
4 |
4.01 |
69.98 |
91.37 |
25646 |
14170 |
279.1 |
366.11 |
6456.1 |
|
5 |
5 |
84.6 |
8 |
3393.1 |
2293.5 |
424.95 |
39.97 |
680.62 |
|
6 |
6 |
97.13 |
72.52 |
42348 |
16152 |
579.01 |
438.73 |
7041.4 |
|
7 |
7 |
45.9 |
45 |
14575 |
5384.1 |
322.41 |
312.98 |
2068.6 |
|
8 |
8 |
19 |
75.4 |
11367 |
4385.5 |
151.79 |
598.06 |
1426.4 |
|
9 |
9 |
13 |
85.9 |
10067 |
4039.8 |
117.19 |
772.99 |
1118.6 |
|
10 |
10 |
58.3 |
38.8 |
22662 |
6462.6 |
579.42 |
388.69 |
2258 |
|
11 |
11 |
13 |
70.7 |
10149 |
3696.6 |
143.22 |
781.77 |
919.96 |
|
12 |
11.97 |
1 |
58.9 |
709.45 |
1552.6 |
12.1 |
709.36 |
59.02 |
|
13 |
12.97 |
41.25 |
72.72 |
39012 |
8967.7 |
531.59 |
948.95 |
2988.6 |
|
14 |
14 |
32.1 |
20.1 |
8984.1 |
2716 |
447.23 |
281.37 |
639.43 |
|
15 |
14.9 |
81.7 |
33.82 |
41533 |
9073.9 |
1231.1 |
510.03 |
2781.9 |
|
16 |
15.9 |
19 |
29 |
8777.3 |
2627.6 |
304.91 |
463.44 |
552.08 |
|
17 |
16.99 |
96.87 |
56.98 |
93719 |
16390 |
1654.3 |
966.7 |
5533.3 |
|
18 |
18 |
72 |
53 |
69195 |
12114 |
1292.9 |
958.57 |
3778.4 |
|
19 |
19.1 |
87.2 |
42.9 |
71005 |
12480 |
1648.7 |
818.62 |
3745.7 |
|
20 |
20 |
79 |
38.1 |
60030 |
10706 |
1586.4 |
762.56 |
3009.8 |
|
21 |
21 |
54.1 |
48.7 |
55591 |
9604.8 |
1135.9 |
1029.1 |
2657 |
|
22 |
22 |
82.2 |
19 |
34111 |
7522.9 |
1811.4 |
415.92 |
1559.2 |
|
23 |
23 |
40.2 |
55.1 |
50306 |
8771.8 |
924.69 |
1267.8 |
2197.8 |
|
24 |
23.9 |
73.9 |
73.1 |
129923 |
17887 |
1779 |
1749 |
5405.9 |
|
25 |
25.1 |
79 |
49.9 |
98788 |
14308 |
1974.1 |
1244.6 |
3962.8 |
|
26 |
26 |
79.7 |
40.1 |
83391 |
12635 |
2070.5 |
1036.8 |
3211.4 |
|
27 |
26.9 |
46 |
2 |
2487.2 |
2779.6 |
1247.8 |
54 |
92.1 |
|
28 |
28 |
53 |
42 |
62189 |
9783.6 |
1486.2 |
1181.6 |
2241 |
|
29 |
29.1 |
32.1 |
52 |
48267 |
8241.3 |
930.8 |
1501.2 |
1664.9 |
|
30 |
30.2 |
35.9 |
79.9 |
86536 |
12669 |
1082.4 |
2403.1 |
2881.8 |
|
31 |
31.1 |
77.9 |
79 |
191451 |
22190 |
2423.5 |
2459.8 |
6181.2 |
|
32 |
31.9 |
64.3 |
68.1 |
139557 |
17054 |
2046.2 |
2168.3 |
4364.9 |
|
33 |
32.9 |
44 |
12 |
17365 |
4729 |
1446.9 |
396.48 |
527.39 |
|
34 |
34.03 |
59.94 |
67.01 |
136942 |
16665 |
2036.4 |
2278.9 |
4025.5 |
|
35 |
35 |
6 |
91.1 |
19030 |
7872.3 |
209.84 |
3181.1 |
545.4 |
|
36 |
36.05 |
57.98 |
25.91 |
54349 |
9032.8 |
2097.8 |
935.39 |
1507.1 |
|
37 |
36.9 |
91.4 |
26.9 |
92248 |
13693 |
3429.6 |
998.3 |
2484.1 |
|
38 |
37.7 |
16 |
25 |
15150 |
3914.6 |
610.5 |
955.26 |
402.52 |
|
39 |
38.98 |
71.67 |
9.02 |
25344 |
7631.4 |
2796.2 |
350.83 |
648.09 |
|
40 |
39.93 |
67.26 |
48.13 |
128831 |
15633 |
2688.6 |
1931.9 |
3215.3 |
|
41 |
40.8 |
51.7 |
31.2 |
66014 |
9995.4 |
2143.4 |
1272.6 |
1611.4 |
|
42 |
41.8 |
33.9 |
46.2 |
65479 |
9891.6 |
1430 |
1929.4 |
1577 |
|
43 |
42.8 |
15 |
6 |
3866.5 |
1995.4 |
646.66 |
256.75 |
89.37 |
|
44 |
43.69 |
75.92 |
14.09 |
47055 |
10072 |
3338.2 |
614.08 |
1061.2 |
|
45 |
45.1 |
95.2 |
100.36 |
427569 |
36505 |
4260.7 |
4503.1 |
9512.1 |
|
46 |
46 |
97.6 |
74.8 |
338064 |
30459 |
4504.3 |
3465.3 |
7352.1 |
|
47 |
47 |
75 |
39.9 |
140990 |
16757 |
3515.4 |
1883 |
2993.1 |
|
48 |
48.2 |
24.8 |
21 |
25168 |
5468 |
1210.2 |
1014.2 |
522.96 |
|
49 |
48.87 |
47.03 |
38.16 |
87468 |
11923 |
2299.2 |
1871 |
1777.6 |
|
50 |
49.5 |
90.7 |
12 |
54845 |
12492 |
4546.9 |
598.39 |
1092.2 |
|
51 |
50.7 |
83.6 |
84.9 |
365753 |
31525 |
4283.4 |
4325 |
7142.9 |
|
52 |
52 |
49.1 |
54.2 |
138096 |
15947 |
2540.9 |
2806.3 |
2648.4 |
|
53 |
52.9 |
91.6 |
19.9 |
96975 |
15529 |
4887.4 |
1062.4 |
1838.1 |
|
54 |
54 |
43 |
8 |
18639 |
6205.5 |
2318.1 |
433.41 |
345.32 |
|
55 |
55.11 |
95.44 |
68.29 |
359308 |
31104 |
5283.9 |
3745.5 |
6532.2 |
|
56 |
56.2 |
86.6 |
64.4 |
312059 |
27985 |
4873.1 |
3589.4 |
5587.8 |
|
57 |
57.1 |
64.1 |
67.2 |
244645 |
23553 |
3646.7 |
3821.6 |
4279.6 |
|
58 |
57.8 |
98.1 |
84.2 |
475935 |
37706 |
5727.3 |
4844.4 |
8271.2 |
|
59 |
59.3 |
90.2 |
25 |
132362 |
18095 |
5295.2 |
1476.3 |
2243.8 |
|
60 |
60.1 |
9 |
12.1 |
6463 |
2739.8 |
542 |
718.57 |
107.12 |
|
61 |
61.17 |
35.99 |
63.98 |
140622 |
16952 |
2186.4 |
3903.6 |
2296.5 |
|
62 |
62.2 |
55.2 |
25.9 |
88211 |
12882 |
3433.3 |
1613.5 |
1419.3 |
|
63 |
63 |
83.4 |
22 |
115059 |
16788 |
5239.4 |
1391.5 |
1819.8 |
|
64 |
64 |
59.9 |
89.4 |
341463 |
29893 |
3855.1 |
5687.6 |
5328.1 |
|
65 |
65.1 |
53.8 |
23.1 |
80424 |
12477 |
3515.6 |
1492 |
1241.1 |
|
66 |
65.9 |
35.9 |
38.1 |
90788 |
12528 |
2370 |
2507.6 |
1366.8 |
|
67 |
67.2 |
13 |
20.1 |
17488 |
4926.4 |
872.78 |
1337.1 |
260.27 |
|
68 |
68.06 |
78.08 |
68.41 |
364870 |
30714 |
5282.5 |
4677.5 |
5447.4 |
|
69 |
69 |
80.1 |
84.2 |
464248 |
35996 |
5512.2 |
5791 |
6753.9 |
|
70 |
70.2 |
45.7 |
45.1 |
144287 |
16847 |
3224.4 |
3154.2 |
2058.9 |
|
71 |
70.8 |
38.1 |
82 |
222377 |
23333 |
2693.5 |
5829.9 |
3114 |
|
72 |
72.4 |
4 |
68.9 |
19880 |
11060 |
289.05 |
4994.3 |
275.5 |
|
73 |
73.14 |
51.87 |
38.13 |
144579 |
17101 |
3784 |
2762.6 |
1979.1 |
|
74 |
73.7 |
60.9 |
67.1 |
302865 |
27091 |
4516 |
4945 |
4069.3 |
|
75 |
75.3 |
81.7 |
95.6 |
593395 |
42470 |
6115.9 |
7156.3 |
7870.9 |
|
76 |
75.9 |
7 |
95.5 |
50468 |
16849 |
532.85 |
7200.6 |
665.79 |
|
77 |
76.81 |
78.84 |
45.72 |
280449 |
26490 |
6070.2 |
3531.8 |
3630.3 |
|
78 |
78 |
12 |
21 |
19557 |
5640.3 |
939.57 |
1639.3 |
253.48 |
|
79 |
78.95 |
40.02 |
100.07 |
316446 |
29963 |
3157.6 |
7905.7 |
3986.5 |
|
80 |
79.6 |
51.1 |
99.3 |
408737 |
34388 |
4064.8 |
8024.3 |
5129.6 |
|
81 |
81 |
4 |
23 |
7470.8 |
4552.3 |
323.47 |
1860.5 |
92.2 |
|
82 |
82.2 |
56.1 |
52.2 |
240217 |
23617 |
4606.2 |
4252.9 |
2903.7 |
|
83 |
82.7 |
78.8 |
98.3 |
640215 |
44866 |
6538.9 |
8105.2 |
7726.1 |
|
84 |
83.7 |
75 |
60 |
376680 |
31771 |
6315 |
5038.5 |
4488.7 |
|
85 |
84.8 |
3 |
17 |
4353.7 |
3514 |
255.25 |
1446.7 |
50.8 |
|
86 |
86 |
72.1 |
31.9 |
197609 |
22588 |
6191 |
2751.4 |
2301.2 |
|
87 |
87.2 |
95.9 |
40.9 |
343067 |
31644 |
8338.8 |
3563.9 |
3931.2 |
|
88 |
87.6 |
31.1 |
76.9 |
210247 |
23745 |
2739.6 |
6815.8 |
2395.3 |
|
89 |
89.4 |
64.9 |
13.1 |
75274 |
15629 |
5776.3 |
1160.3 |
846.53 |
|
90 |
90.29 |
41.84 |
92.76 |
352882 |
32026 |
3778.3 |
8414.4 |
3918 |
|
91 |
90.9 |
54 |
69.4 |
339522 |
29850 |
4899.4 |
6260.6 |
3707.8 |
|
92 |
92.2 |
9 |
2 |
1646.4 |
2061.2 |
829.44 |
184.08 |
18.02 |
|
93 |
93.4 |
53.14 |
98.91 |
487931 |
38467 |
4923.4 |
9204.6 |
5223.8 |
|
94 |
94.1 |
24.1 |
23.2 |
52222 |
9977.7 |
2254.2 |
2163.6 |
552.84 |
|
95 |
94.58 |
47 |
24.99 |
111323 |
16045 |
4450.6 |
2361.8 |
1176.1 |
|
96 |
96.2 |
1 |
59.3 |
5687.1 |
11636 |
96 |
5645.6 |
59.1 |
|
97 |
96.3 |
96.7 |
85.2 |
793775 |
51747 |
9364.4 |
8316.5 |
8259.3 |
|
98 |
97.9 |
92.1 |
65.1 |
581293 |
42819 |
8993.7 |
6365.1 |
5975.7 |
|
99 |
98.4 |
26.9 |
72.1 |
192615 |
23405 |
2672.5 |
7160.2 |
1947.3 |
|
100 |
99.9 |
47.2 |
83.9 |
393740 |
34157 |
4703.5 |
8371.5 |
3957.8 |